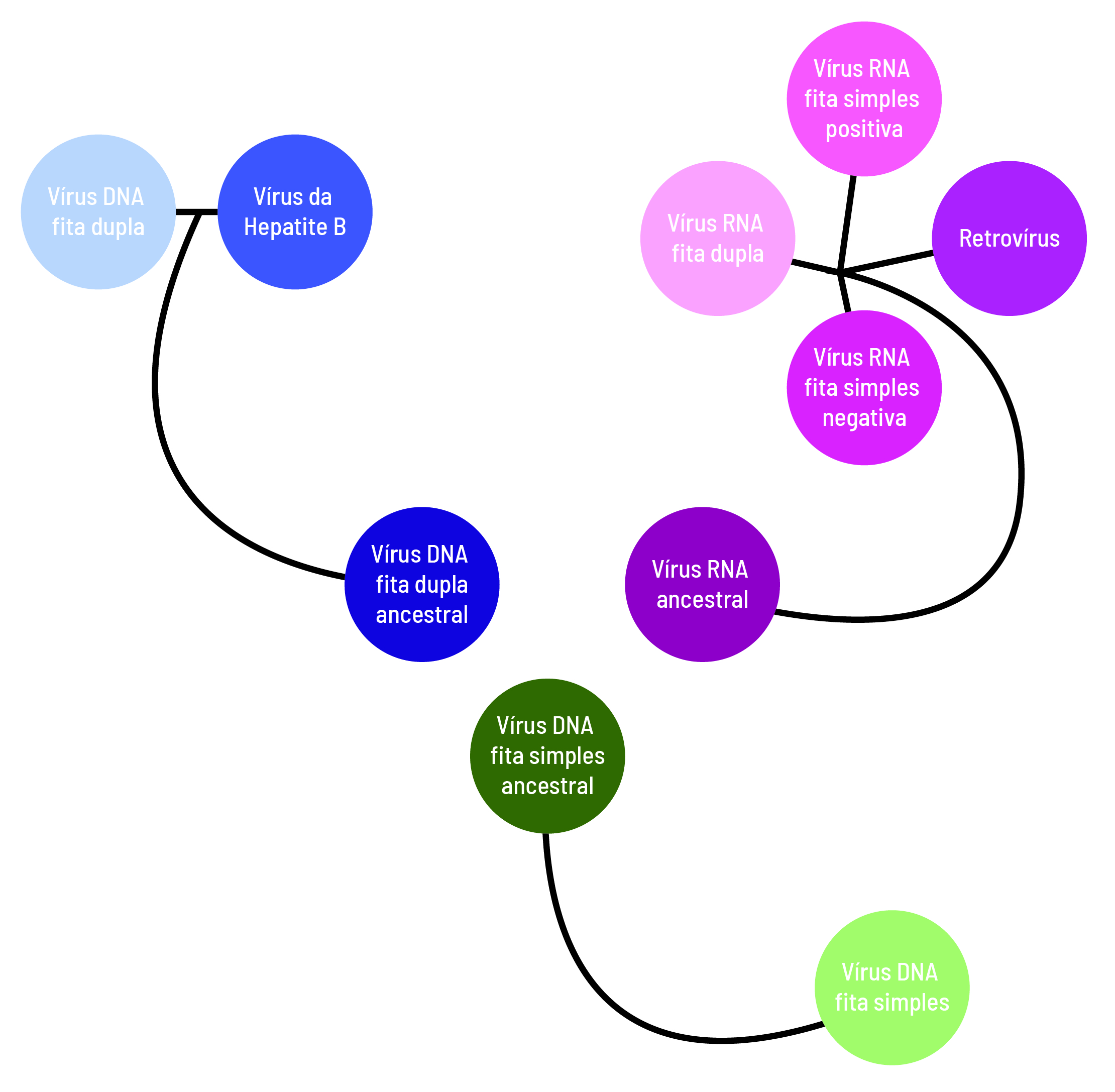
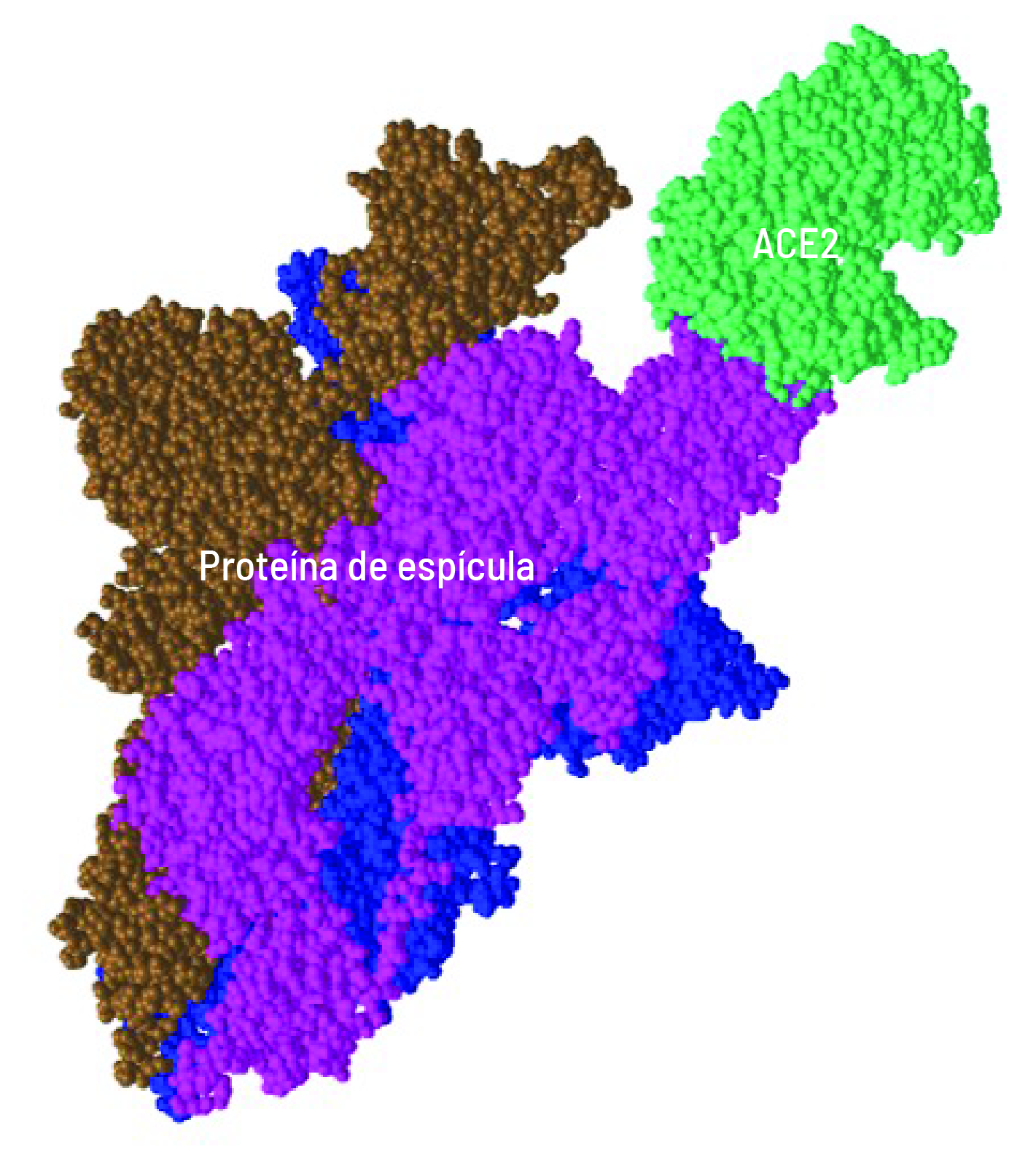
Vivemos sob o impacto mundial das diferentes variantes do coronavírus que causa a covid-19. Embora contemos com tecnologia e conhecimento para que uma nova pandemia não nos pegue de surpresa, é preciso um investimento adicional: dirigir os esforços para a pesquisa da origem e da evolução desses vírus. Só assim aprimoraremos nosso entendimento sobre o comportamento e a diversidade das espécies para um combate mais eficaz e específico.
CRÉDITO: A PARTIR DE FOTO ADOBE STOCK